ゲノム編集に汎用されるCRISPR-Cas9を改変
広島大学は10月17日、ゲノム編集に汎用されるCRISPR-Cas9を改変し、DNA配列を書き換えることなく遺伝子の働きをONにする遺伝子の活性化技術「TREEシステム」を開発したと発表した。この研究は、同大大学院理学研究科の佐久間哲史講師、山本卓教授らが、国立がん研究センター研究所エピゲノム解析分野の牛島俊和分野長、川崎医科大学総合外科学講座の深澤拓也准教授らと共同で行ったもの。研究成果は「The CRISPR Journal」に掲載された。
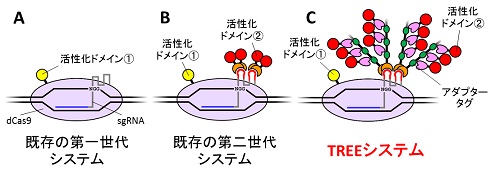
遺伝子を活性化させるシステムとして、これまでにDNA結合タンパク質に活性化ドメインを直接連結させた第一世代の構造や、dCas9と複合体を形成するsgRNAに活性化ドメインを呼び込む第二世代の構造などが報告されてきた。しかし、これらの構造では必ずしも十分な活性化効果が得られるとは限らず、より高い効果を発揮する新規システムが望まれていた。
膵臓がん細胞でE-カドヘリンタンパク質の発現レベルを約30倍に
今回の研究では、まずsgRNAにアダプター分子となるマルチタグタンパク質を呼び込み、さらにそのタグの一つひとつに活性化ドメインが結合することで、目的の活性化ドメインが高度に集積し、より強い活性化が誘導されるようなシステムを構築した。このシステムは、「発現増強のための3要素型の別用途化技術」を意味するThree-component Repurposed technology for Enhanced Expressionの略称としてTREEシステムと命名。システムを模式的に示す際にツリー状の形状となることも、名称の一因となったという。
画像はリリースより
TREEシステムの優位性は、複数の細胞株を用いて、複数の遺伝子座で実証された。そのうちのひとつが、MIA-PaCa2とよばれる膵臓がん細胞でのCDH1遺伝子の発現増強だ。CDH1遺伝子は、がん抑制遺伝子で、細胞接着に関わるE-カドヘリンタンパク質をコードしている。MIA-PaCa2細胞において、E-カドヘリンタンパク質の発現量は本来検出限界以下であり、既存の第一世代の活性化システムを作用させた場合でも検出可能な発現は認められなかった。既存の第二世代の活性化システムでは、ごくわずかに発現が検出可能になったが、TREEシステムを用いることで、第二世代システムによる発現レベルの30倍におよぶ活性化効果が実現されたという。
今回開発されたTREEシステムは、がんの発症をモデル化するための技術としても活用されることが期待される。また、再生医療用細胞を作製するためのリプログラミングへの応用も可能と考えられるという。高度遺伝子活性化技術として極めて有望であるだけでなく、さまざまな用途への応用も可能な汎用的基盤技術である、と研究グループは述べている。
▼関連リンク
・広島大学 研究成果



